
- Main
- Files
- Control Panel
- Selecting
- Move & Rotate
- Display
- Rendering
- Tools
- Mutations
- Torsions
- Preferences
- Electrostatics
- Surface
- Scripting
- Hardware stereo
- Tips & Tricks
- Download Manual
Potencjał elektrostatyczny
W tej części dowiemy się, jak obliczyć potencjał elektrostatyczny lub użyć możliwości komputerowych poprzez zastosowanie innych programów.
Potencjał elektrostatyczny białek - spowodowany przez naładowane łańcuchy boczne oraz przez związane jony - odgrywa ważną rolę, np. podczas zwijania białek i ich stabilizacji, katalizy enzymów lub podczas tworzenia wiązań białko-białko. Ogólnie, potencjał elektrostatyczny przedstawiany jest jako:
- Potencjał elektrostatyczny na mapie:
W tym przypadku można zmieniać wartości kształtujące potencjał (w kT/e) w czasie rzeczywistym, poprzez strzałki góra i dół (lub przez otwarcie okna EDM i kliknięcie na wartość sigma). Pozytywne potencjały są rysowane w kolorze niebieskim, negatywne na czerwono, ale te kolory mogą być zmienione przez kliknięcie w polu "color box" w oknie EDM.
E.g. rozmieszczenie ładunków w bydlęcej katalizy alfa (catalytic alpha) składa się z proteiny cAMP zależnej Kinazy (1YDR)która formuje charakterystyczne anizotropowe pole elektryczne produkowane do otaczającego rozpuszczalnika (wyświetlane jako siatka lub p {??}) to ustawien ie może być zmodyfikowane w preferencjach EMAP:


W tym obrazie, włączony jest widok wstążki, a wszystkie pozostałe struktury drugorzędowe zostały wybrane jako "zwój" z menu "Edit" celu uzyskania struktury robaka.
- albo mapowany na powierzchni molekularnej
Aby obliczyć powierzchnie molekularne potencjału elektrostatycznego, należy włączyć opcję "color by electrostatic potential" w preferencjach "Surface" i obliczyć powierzchnię (pamiętaj, że możesz również załadować powierzchnię przy użyciu innych programów opisanych poniżej). Jeżeli zaznaczymy opcje "Map potential to surface" w menu preferencji "Electrostatics" to nowe potencjały elektrostatyczne, zostaną mapowane do istniejącej powierzchni, kiedy zostaną one obliczone lub załadowane. Ten typ przedstawień powierzchni jest przydatny np. do porównania interfejsów protein.
Ta sama cząsteczka jak powyżej, tylko w kompleksie z peptydowym inhibitorem białka kinazy H7, teraz potencjał został obliczony na powierzchni: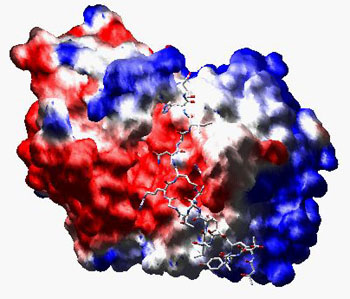
Ustawienia potencjału elektrostatycznego można zmieniać w preferencjach "Electrostatic Potential". Obecnie proteina jest ustawiona na pH=7,0 z domyślnym stanem dla wszystkich reszt. Również domyślnie jedynie naładowane reszty (Arg, Lsy, Glu, Asp) są brane pod uwagę podczas obliczania. Możesz także skorzystać z ładunków częściowych 43A1 GROMOS pola siłowego. To jest znacznie wolniejsze, bardziej naładowane atomy są brane pod uwagę.
Normalne potencjały obliczane są przy użyciu prostych interakcji - kulomb. Jest to najszybsza metoda, ale zarazem mało dokładna, ponieważ jest stosowana tylko jednolita stała dielektryczna, stosuje się ją do wnętrza białka jak i do przestrzeni rozpuszczalnika. Jeśli chcemy uwzględnić zależności dielektryczne wnętrza białka i rozpuszczalnika musimy wziąć pod uwagę równanie numeryczne Poissona-Boltzmanna (ta procedura nie została jeszcze wdrożona w SPDV, ale dzięki niej można wyświetlić potencjał PB obliczony przy pomocy DELPHI).
SPDBV pozwala również na wizualizację powierzchni lub potencjałów obliczonych przez inne programy:
- DELPHI - mapy potencjałów
Program DELPHI oblicza rozwiązanie numeryczne z równania PB. [Honig and Nicholls, Science (1995) 268, 1144-1149] - GRASP - powierzchnie
GRASP oblicza powierzchnie molekularne i proste rozwiązania numeryczne PB na ograniczonej sieci. SPDBV odczytuje pliki w formacie zwykłej powierzchni (fort.25, *. srf)z lub bez elektrostatycznych potencjałów [Ref: Anthony Nicholls, Kim Sharp and Barry Honig; Proteins (1991) vol. 11 pp 281].
Pamiętaj, że możesz również uzyskać dostęp do strony internetowej, dzięki której można obliczyć powierzchnie oraz różne właściwości map na nich. (from Barry Honig's lab.) - MSMS
- powierzchnie molekuł
Program MSMS oblicza analityczne powierzchnie molekularne. [Ref: Michael F. Sanner, Olson & Spehner, Biopolymers (1996) 38, 305]